Q Q:800 176 181
电话:400-167-8986
手机1:185 1867 6727
手机2:173 0710 7886
订货:order@genenode.com
技术:service@genenode.com
网址:www.genenode.com
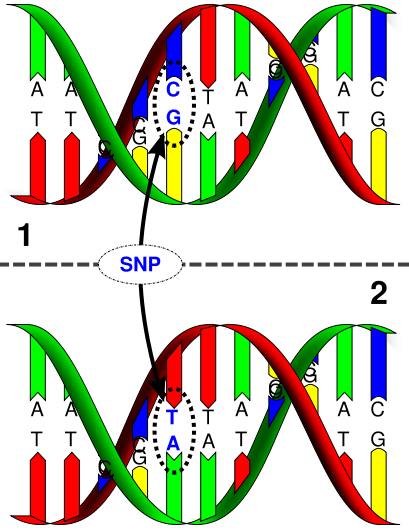
SNP|单核苷酸多态性分析|SNP基因分型
SNP/单核苷酸多态性分析技术平台: 1.基因突变检测(PCR-测序法) 2.Taqman-MGB探针SNP分型 3.PCR-RFLP酶切SNP分型 4.PCR-LDR连接酶检测SNP分型 5.SNaPshot法SNP分型 6.Sequenom MassArray SNP分型 7. KASP SNP分型
单核苷酸多态性(Single Nucleotide Polymorphisms,SNPs),指由于单个核苷酸碱基的改变而导致的核酸序列的多态性。在不同个体的同一条染色体或同一位点的核苷酸序列中,绝大多数核苷酸序列一致而只有一个碱基不同的现象,这就是SNP。
由于SNP在人类基因组中数量较多,发生频率较高,因此被认为是继微卫星之后的新一代遗传学标记,在医学遗传学、药物遗传学、疾病遗传学、疾病诊断学、以及人类进化等研究领域都有着很高的研究价值和应用前景。
订单点击下载:SNP基因分型订单表
SNP/单核苷酸多态性分析技术平台:
1. 直接测序法(PCR-测序法)
2.Taqman-MGB探针SNP分型
3.PCR-RFLP酶切SNP分型
4.PCR-LDR连接酶检测SNP分型
5.Multiplex SNaPshot SNP分型
6.Sequenom MassArray SNP分型
7. KASP SNP分型
1.1 技术原理:对欲检测片段进行直接PCR扩增、采用ABI 3730xl DNA测序仪测序,纯合型SNP位点的测序峰为单一峰型,而杂合型SNP位点的测序峰为套峰,因而很容易将其区分开来,通过直接测序方法进行SNP检测的检出率接近100%。测序分型主要通过直接测序的方法来确定位点的基因型,这种分型方法准确性最高,但费用大。如果需要检测的几个SNP位点正好位于一个测序单元内(长度小于700bp),则单个位点的分型费用可显著降低,这种分型技术不失为一种良好选择。对于准确性要求特别高或样本量特别小(几个或几十个)的项目,这种分型技术非常适合。
实验结果图:
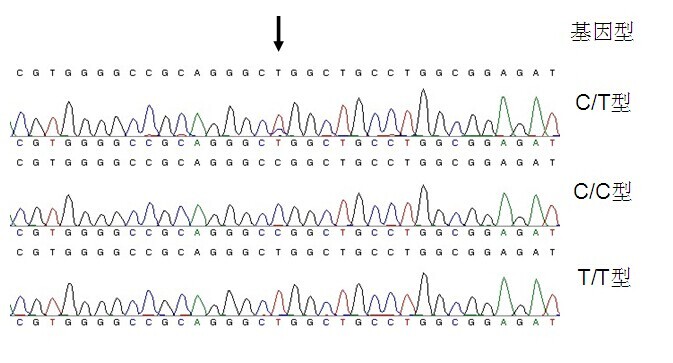
1.2 服务内容:
1. 样品基因组DNA提取: 需收取核酸提取费用
2. 引物设计及合成: 仅收取引物合成费用
3. PCR扩增及纯化: 需收取PCR扩增及纯化费用
4. ABI 3730xl测序: 测序费用
5. 数据分析整理: 测序峰图分析,SNPs结果和实验报告整理
1.3 适宜范围:
1. 需要检测的几个SNP位点正好位于一个测序单元内(长度小于600bp)的分型项目;
2. 对于准确性要求特别高或样本量特别小(几十个)的分型项目,如对其它SNP分型方法准确性的验证。
2.1 技术原理:以SNP分型检测为例,在PCR过程中,两个探针能与正向引物和反向引物之间的互补序列特异退火结合。当探针以完整形式存在时,由于能量共振转移,荧光基团只发出微弱荧光。特异的探针与相应的等位基因杂交后,DNA聚合酶发挥5’外切酶活性,把报告荧光基团切割下来,从而发出荧光;而SNP探针的错配形式不会引起切割,无荧光信号。MGB分子结合到DNA螺旋小沟,通过稳定MGB探针/模板提高杂交稳定性,使短至13个碱基的探针获得高错配区分辩别能力。MGB探针包括一个不发荧光的淬灭基团(NFQ),极大消除传统淬灭基团产生的背景荧光,提高信噪比,从而提高检验灵敏性。
原理及流程示意图:
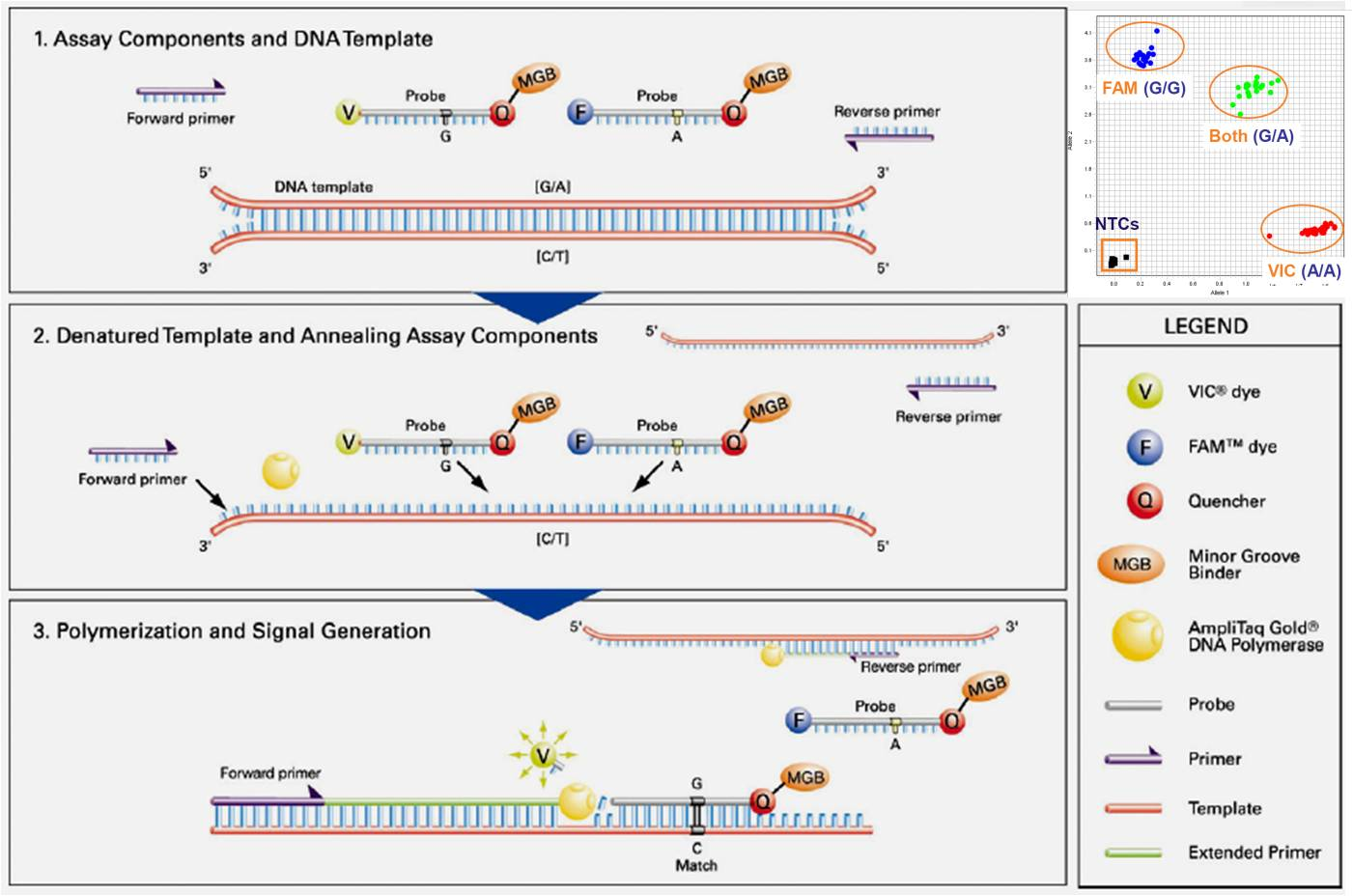
2.2 技术优势:
1. 采用最先进的ABI 7900HT系统采用激光扫描并激活96/384孔板每个反应孔的荧光染料,荧光信号通过光栅分光,经过分离的多色荧光同时到达CCD摄像机,实现多色荧光同时检测。
2. 采用ABI 专利技术的TaqMan-MGB 探针:MGB探针包括一个不发荧光的淬灭基团(NFQ),极大消除传统淬灭基团产生的背景荧光,提高信噪比,从而提高检验灵敏性,另外MGB(小沟结合物)增加Tm值,使探针设计产度缩短至约15bp,提高了探针设计的灵活性。
2.3 技术特点:
1. 适合位点少,样本通量高的检测,每个位点需要合成一对探针,探针合成费用高;
2. 应用ABI7900、ABI7500定量PCR仪进行定量检测;
3. 操作简便,只需一步 PCR 反应,无须纯化和预处理,直接上机检测;
4. 结果清晰,软件分析结果界面友好,标出每个样本的基因型及荧光强度,非常直观;
2.4 适用范围:
特别适合样本数量超过1500个以上,位点数量比较少的SNP分型。
3.限制性片段长度多态性聚合酶链反应(PCR-RFLP)技术
3.1 技术原理:
用PCR扩增目的DNA,扩增产物再用特异性内切酶消化切割成不同大小片段,由于不同等位基因的限制性酶切位点分布不同,产生不同长度的DNA片段条带,所以可以直接在凝胶电泳上对其多态性进行分辨。
原理及流程示意图:
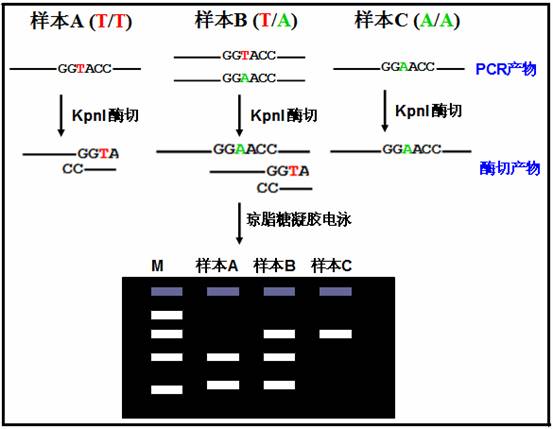
3.2 技术特点:
1. 适合位点数量少,样本通量高的检测(检测位点需要有合适的酶切位点存在)
2. 结果清晰直观
3.3 服务内容:
1. 位点查询,内切酶确定购买
2. 扩增引物设计,调试
3. PCR扩增特定位点,PCR产物电泳检测
4. 酶切鉴定分析,电泳检测
5. 电泳检测结果记录,数据统计分析
4.1 技术原理:
主要应用连接酶检测反应 (ligase detection reaction,LDR)技术。即在Taq DNA连接酶的作用下, 当左右两条寡核苷酸探针与目的DNA序列完全互补,并且两条探针之间没有空隙时才能发生连接反应,否则连接反应不能进行,最后通过荧光扫描片段长度,根据测序峰移动的胶位置,通过末端未标记探针的3个碱基差异,确定同一位点的不同基因型,再通过末端未标记探针的7个碱基差异,确定不同位点,最终实现对SNP 位点的精确检测。
原理及流程示意图:
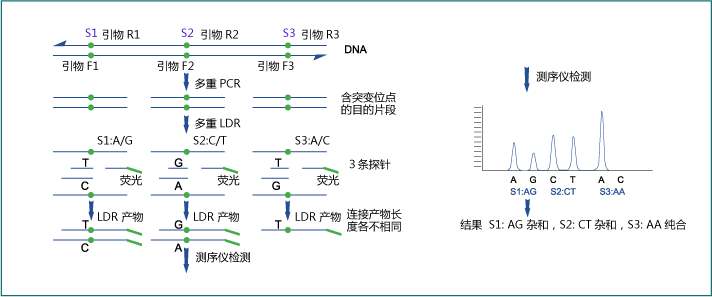
4.2 PCR-LDR技术流程:
1. 多重PCR扩增:获得目标位点所在的片断;
2. 多重LDR:得到长度各异的检测产物;
3. 3730测序仪检测:根据测序电泳得到不同的峰值;
4. 判断SNPs结果,数据分析;
4.3 方法特点:
1. 通用性强:适用任何多态性位点和各种SNP位点的分型,不需要人工引入酶切位点;
2. 分型准确,检出率高:其准确度可达99.2%以上。
3. 检测快速:LDR技术的最大优点就是实验时间短!与Taqman、RFLP、SSCP和DHPLC相比,由于检测条件更易控制,从DNA样本到数据的产出,整体实验只需要几周。
4.4 适宜范围:
适宜 100-1000个样品,1-5个位点;
5.1 技术原理:
在一个含有测序酶,四种荧光标记的ddNTP,紧挨多态位点5’端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪跑胶后,根据峰的颜色可知掺入的碱基种类,从而确定该样本的基因型,根据峰移动的胶位置确定该延伸产物对应的SNP位点。对于PCR产物模板可通过多重PCR反应体系来获得,通常用于6-30个SNP位点分析。
技术原理:

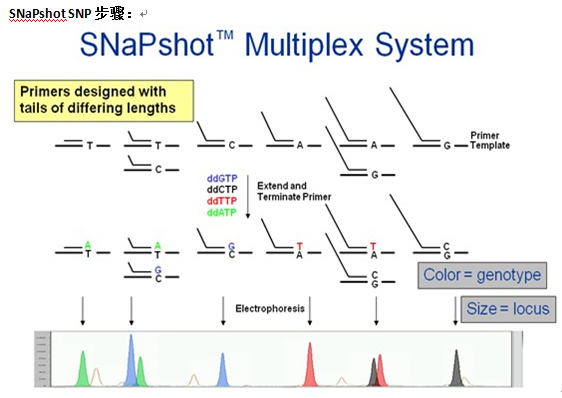
5.2 技术优势:
1. 分型准确:该技术也称微测序技术,其准确度仅亚于直接测序;
2. 多位点同时检测:最多可同时检测12个位点;
3. 可检测出受污染的样本:如果一个样本的分型峰谱偏离正常的分布,它可提示该样本可能受到污染或浓度过低;
5.3 适宜范围:
适合中高通量SNP检测,位点范围6-50个,样本量范围100-3000个。
6.1 技术原理:先通过PCR扩增目标序列,然后加入SNP序列特异延伸引物,在 SNP位点上,延伸 1个碱基。将制备的样品分析物与芯片基质共结晶后在质谱仪的真空管经强激光激发,核酸分子离解吸附为单电荷离子,电场中离子飞行时间与离子质量成反比,通过检测核酸分子在真空管中的飞行时间而获得样品分析物的精确分子量,从而检测出SNP位点信息。
原理及流程示意图:
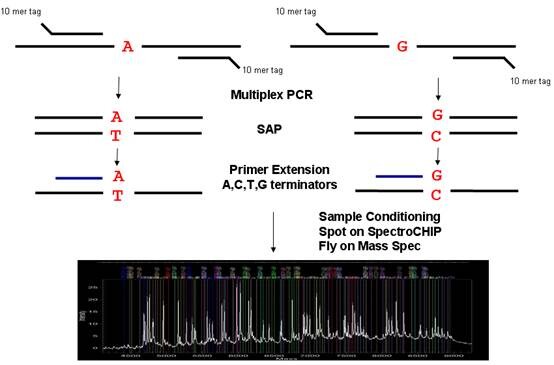
6.2 技术优势:
1. 高通量检测,简单灵活:采用Sequenom 384芯片平台,能为受检样品设计最高可达30重的PCR反应,从而通过一次实验即可获得每个样品多达30个SNP位点的多态性研究数据,节省检测经费,缩短检测时间,提高了检测效率。用户可以自行定义待测SNP位点和样品数,每天能够检测6张384芯片/台仪器,单个样品一次最多可检测30多个位点,位点检出率≥90%,样品检出率>95%,重复性>98%;
2. 结果准确,灵敏度高:结合单碱基延伸技术,利用质谱直接检测物质分子量,MassArray反应体系为非杂交依赖性,不存在潜在的杂交错配干扰,也不需要引入各种荧光标记物,有效避免由于荧光标记效率导致的实验误差,实验检测结果准确,稳定性强,结果重复性好;
6.3 适宜范围:适合中高通量SNP检测,简单灵活,结果准确,灵敏度高,价格实惠;
7.1 技术原理:
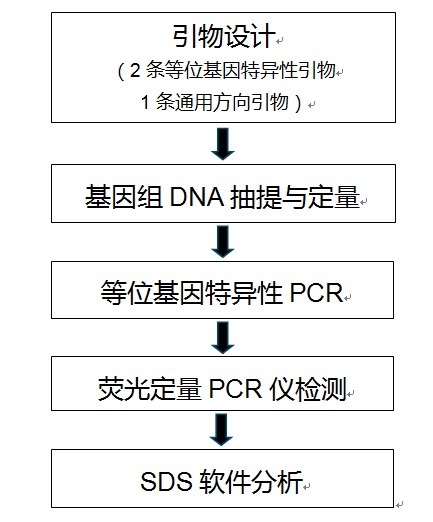
KASP-竞争性等位基因特异性PCR(Kompetitive Allele-Specific PCR),该技术是基于引物末端碱基的特异匹配来对SNP分型以及检测InDels (Insertions and Deletions,插入和缺失),采用2个荧光探针、2个通用淬灭探针,再加多个位点特异探针来检测多个SNP位点,采用全自动核酸提取仪、荧光定量PCR平台,灵活采用96孔板、384孔板,一天可检测几万个SNP 位点。
KASP技术原理上类似Taqman检测,也是基于终端荧光读取判断,每孔采用双色荧光检测一个样本的一个位点可能的两种基因型,而KASP技术与Taqman技术不同的是,它不需要每个SNP位点都去合成特异的荧光引物,它基于自己独特的ARM PCR原理,让所有的位点检测最终都使用通用荧光引物扩增,这大大降低了LGC KASP的试剂成本,既有金标准的准确,又降低了使用成本,比Taqman还具有更好的位点适应性,所以在医学、农学检测方面KASP都有非常好的应用。
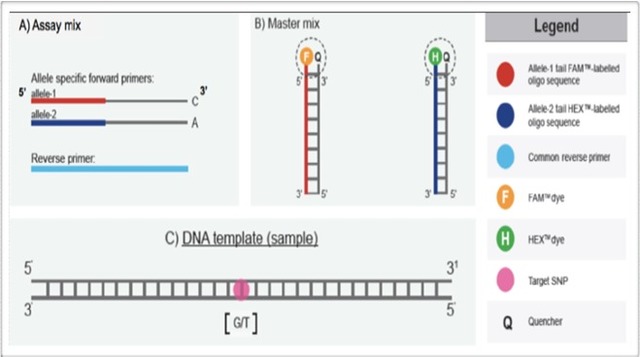
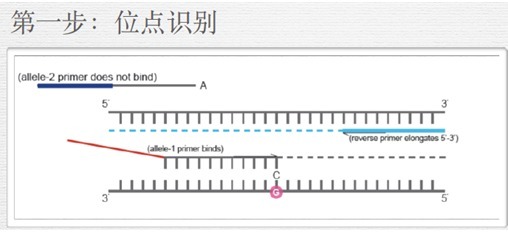
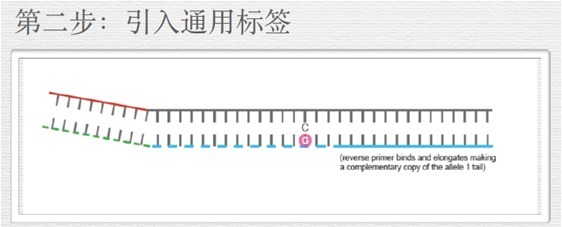
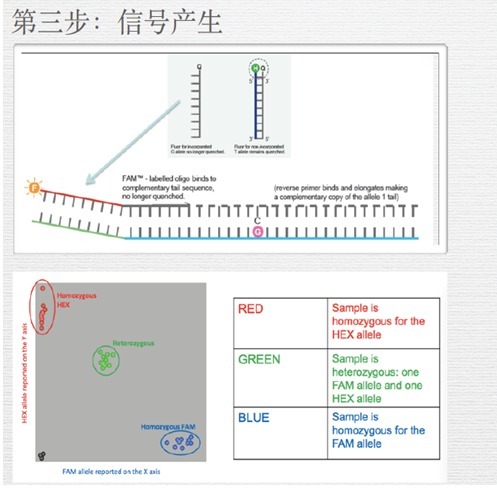
7.2 KASP技术特点:
1.优异的准确性
- 独立评价的准确性>99.8%
- 业内领先的SNP&InDel 分析的转化率(>90%)
2.超强的灵活性
- 设计的高度灵活性带来更高的成功率
- 支持低/中/高通量研究以及单个重复实验
- 对液体处理系统,PCR仪和荧光分析仪等设备有良好的兼容性
3.前所未有的低成本
- 无需昂贵的双色标记探针,同样成本获得两倍数据量
- 最低的DNA样本需求 (根据基因组大小不同只需 0.1-10 ng)
- 无需全基因组扩增 (Whole Genome Amplification, WGA)
由于KASP实验方案的高度灵活性,其应用也十分广泛,适合于各种基因分型研究,无论是中低通量SNP研发项目,NGS之后的SNP验证,农业种群研究,或者大量种子的生产QC和生物样品库 DNA Biobanking等都是其适用的范围。
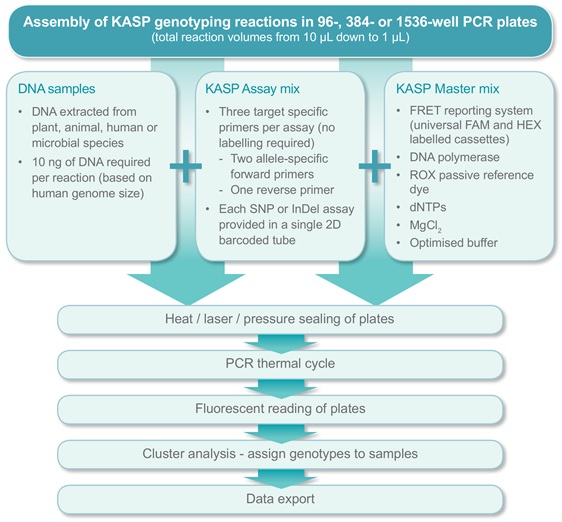
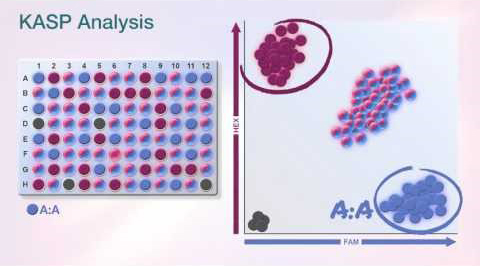
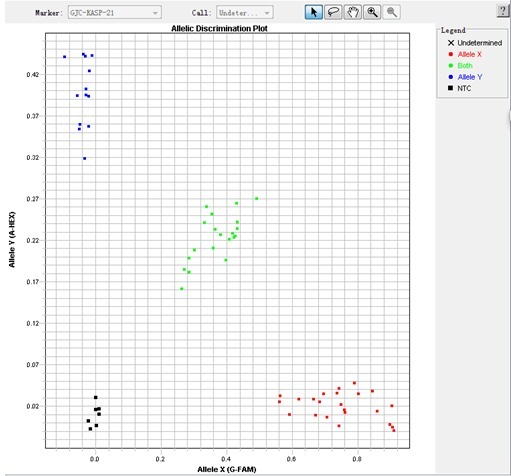
实验步骤:
散点图结果:
服务内容:根据客户的要求和课题特点,提供一站式SNP基因分型服务
1、课题的设计咨询包括检测位点的筛选;
2、全血和组织样本基因组DNA提取(费用另算);
3、DNA样本的质检;
4、根据样品数量和分型位点数,推荐成本最低的分型方法;
5、SNP分型所用引物的设计、合成与优化;
6、SNP基因分型;
7、分型数据的质量质控;
8、提交实验报告
分型数据的质量质控:
1、阴性阳性对照:每批分型实验都设计阴性(H2O)、阳性对照;
2、重复对照:在实验完成后随机选取5%的样品做重复实验(单独收费);
3、测序验证:随机选取2%的样品进行测序验证(单独收费)。
客户需要提供:
1. 细胞(≥106 个)、组织(≥300mg)、血液(≥1ml)、血清(≥1.5ml)等样品材料,核酸提取费用单独收费;
2. 基因组DNA(体积≥30μl,浓度≥50 ng/μl),纯度 OD260/280 为1.7~1.9之间;
3. 人类基因组需提供SNP位点的RS号。其它物种如牛、鸡、鱼等无rs号的,需提供SNP位点上下游各200bp的确切序列,SNP位点的突变类型,以及位点的上下游25bp内是否存在其它连锁位点。
最终提交结果:
1. SNPs结果(ExceL表格);
2. 完整实验报告:扩增和反应体系、所涉及的引物、探针序列;
3. 客户所需要的其他相关资料;


官方微信
联系电话
18518676727
即时通讯
QQ:800176181/1195537948
电子邮箱
order@genenode.com
联系地址
湖北省武汉市长江新区余泊北路99号
阳逻港华中国际产业园C区C5-1
武汉君诺德生物技术有限公司 版权所有 鄂ICP备2021022499号-1 网站地图 网站xml